Espèce de… quoi d’ailleurs ?! Une solution pour le savoir : le barcoding !
Les taxonomistes et les écologistes sont souvent confrontés à des problèmes d’identification d’espèces pour diverses raisons : caractères morphologiques complexes, clés d’identification imprécises, diminution du nombre d’experts en taxonomie, diversité cryptique (non basé sur des caractères morphologiques),… Depuis quelques années, le barcoding moléculaire révolutionne l’approche taxonomique et l’identification d’espèces. Cette stratégie, basée sur la reconnaissance d’un fragment cible de l’ADN spécifique d’une espèce, permet d’identifier les espèces alors même que les espèces sont difficilement ou ne sont pas identifiables d’un point de vu morphologique (fragment d’individu, graine, etc.).
A la base du barcoding, le séquençage de l’ADN
 La biologie moléculaire a commencé à se développer à partir de la découverte de la structure de l’ADN en double hélice en 1953 (Watson and Crick, 1953). Depuis, cette discipline n’a cessé de connaitre de nouvelles avancées.
La biologie moléculaire a commencé à se développer à partir de la découverte de la structure de l’ADN en double hélice en 1953 (Watson and Crick, 1953). Depuis, cette discipline n’a cessé de connaitre de nouvelles avancées.
En 1977, deux techniques concurrentes sont mises au point pour séquencer l’ADN, c’est-à-dire déterminer l’enchaînement des bases qui le compose : celle de Maxam et Gilbert, d’un part, qui est une méthode chimique de coupure des bases, et celle de Sanger, d’autre part, qui utilise un procédé requérant une enzyme : l’ADN polymérase. Ces deux techniques via l’utilisation d’électrophorèse ultra-résolutive permettent la séparation de molécules d’ADN ne différant que d’un seul nucléotide. La méthode de Sanger est aujourd’hui la plus largement utilisée (Lionnet and Croquette, 2005).
Les avancées technologiques de ces vingt dernières années ont permis d’automatiser le séquençage de l’ADN, rendant ce dernier rapide et relativement peu onéreux. De ce fait, une quantité considérable de données génomiques ont été et continue d’être générées. Elles sont souvent regroupées dans des bases de données publiques accessibles à l’ensemble de la communauté scientifique.
Le séquençage de l’ADN permet de caractériser génétiquement des individus.
Principe du barcoding moléculaire
Le barcoding moléculaire, principe relativement récent proposé en 2003 par Paul Hebert, est une extension de cette technique de séquençage ADN.
Il s’agit d’un outil taxonomique permettant la caractérisation génétique d’un individu ou d’un échantillon d’individu à partir d’un fragment standard de l’ADN. En effet, certains fragments d’ADN sont très conservés au sein d’une même espèce mais variables entre les espèces. Les séquences de ces fragments d’ADN appelés barcodes ou code-barre à ADN sont compilées dans des bases de données. Une fois un échantillon inconnu séquencé, il suffit de comparer la séquence ADN trouvée à celles déjà présentent dans les bases de données pour identifier le nom de l’espèce associée à cette séquence (BOLD ou GenBank). Il est ainsi facile et rapide de savoir à quelle espèce appartient l’individu analysé à l’instar des codes-barres sur les produits commerciaux (Hebert et al., 2003). La seule limite à cette approche est d’avoir accès à des bases de données contenant des séquences fiables et validées par la communauté scientifique, ce qui n’est pas toujours le cas.
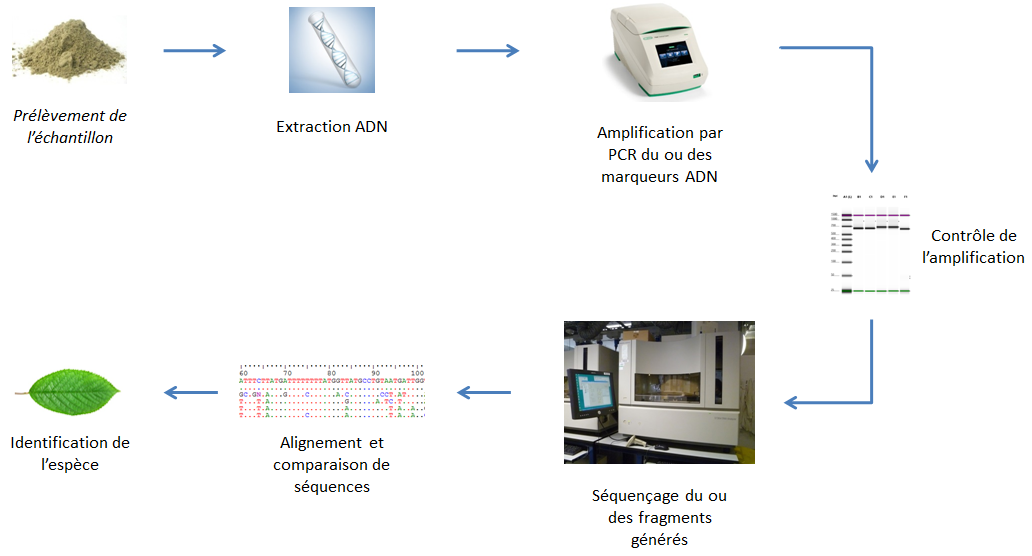
Étapes d’une analyse par barcoding
Le fragment d’ADN à séquencer choisi dans la majorité des cas est un morceau du gène mitochondrial COI (codant pour une sous-unité de l’enzyme cytochrome oxydase, impliqué dans la chaîne de réactions biochimiques de la respiration). Ce gène a l’avantage d’être présent en grande quantité (dans chaque mitochondrie) et d’être assez similaire chez les individus d’une même espèce, alors qu’il montre une forte variabilité interspécifique. Il est apparu cependant que l’utilisation de ce seul marqueur n’était pas appropriée pour tous les taxons. Pour certaines familles taxonomiques, en plus du marqueur COI, d’autres gènes (mitochondriaux ou chloroplastiques) sont aujourd’hui utilisés comme codes-barres.
Applications et utilisations du barcoding
 Aujourd’hui, les applications du barcoding, en agriculture et dans l’industrie sont multiples et ne cessent d’évoluer. Cette approche permet notamment :
Aujourd’hui, les applications du barcoding, en agriculture et dans l’industrie sont multiples et ne cessent d’évoluer. Cette approche permet notamment :
- De détecter la présence ou l’absence de micro-organismes bénéfiques et pathogènes dans l’environnement qui seront favorables ou défavorables aux cultures et pouvoir ainsi adapter les itinéraires techniques en conséquence ;
- De contrôler la conformité et l’absence de contaminations des matières premières utilisées par les industriels (pharmacologie, cosmétique, agro-alimentaire,…) (voir notre billet sur Identification d’espèces d’algues : barcoding !) ;
- De surveiller le commerce illégal de ressources naturelles en analysant des échantillons de viande ou d’écorces dans les marchés locaux ou dans des cargaisons à destination de l’export ;
- De contrôler la qualité de l’eau en disposant d’une connaissance fine de la microfaune et microflore d’un lac, d’une rivière.
Pour aller plus loin, il existe une technique dérivée du barcoding qui permet d’étudier la richesse taxonomique d’un environnement ou réaliser une analyse différentielle de la structure des communautés microbiennes entre deux échantillons, par exemple de sol. Cette technique appelée métabarcoding permet grâce aux nouvelles méthodes de séquençage massif (NGS, Ilumina MiSeq 2×250) l’identification de toutes les espèces présentes dans un échantillon. Sur l’étude de la diversité microbienne globale d’un environnement, retrouvez notre précédent billet Métagénomique : la face cachée de l’iceberg microbien.
Références :
- Hebert, P.D.N., Cywinska, A., Ball, S.L., and deWaard, J.R. (2003). Biological identifications through DNA barcodes. Proceedings of the Royal Society B: Biological Sciences 270, 313–321.
- Watson, J.D., and Crick, F.H.C. (1953). Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature 171, 737–738.
- Lionnet T., Croquette V. (2005). Introduction à la Biologie Moléculaire.
Ce billet a été rédigé conjointement par :
- Céline Hamon, responsable R&D en biologie moléculaire
- Charlotte Roby, ingénieur en biologie moléculaire
- Juliette Clément, chargée de veille et recherche documentaire
Crédits photos : Bar code. Vector illustration. © vahlamov_anton - Fotolia Red DNA string front view isolated on a white background 3d rendering © juancamilo - Fotolia Green leaf isolated © AlenKadr - Fotolia Fields cultivated in spring © MAURO - Fotolia




