La sélection assistée par marqueurs (SAM)

Ou comment optimiser la sélection de nouvelles variétés de plantes ?
L’objectif de l’amélioration des plantes est de créer de nouvelles variétés combinant un certain nombre de caractères définis par le sélectionneur pour répondre aux besoins des agriculteurs et des consommateurs.
Dans les années 1950, le sélectionneur ne faisait appel qu’aux outils de la sélection « phénotypique » avec croisement, autofécondation et expérimentation au champ. Aujourd’hui, les marqueurs moléculaires et les études de génomique apportant une connaissance de plus en plus précise des gènes qui contrôlent des caractères physiologiques, la sélection se fait majoritairement sur le génotype. On parle alors de « Sélection Assistée par Marqueurs » (SAM).
Qu’est-ce que la SAM ?
La SAM est basée sur l’utilisation de marqueurs moléculaires, de petits segments d’ADN situés près d’un gène d’intérêt (résistance à une maladie, par exemple) dans l’ADN de la plante. Les marqueurs sélectionnés et utilisés au laboratoire permettent de détecter précisément la présence ou l’absence du (ou des) gène(s) d’intérêt recherché(s) dans les plants en cours de sélection.
La SAM présente également un intérêt important dans les programmes d’introgression d’un gène d’intérêt dans une variété élite par rétrocroisements (ou back-cross, Figure 1). Les marqueurs sont dans ce cas utilisés pour suivre l’introgression des allèles favorables au gène d’intérêt et accélérer le retour au fonds génétique élite souhaité (en utilisant des marqueurs moléculaires neutres répartis sur tout le génome).
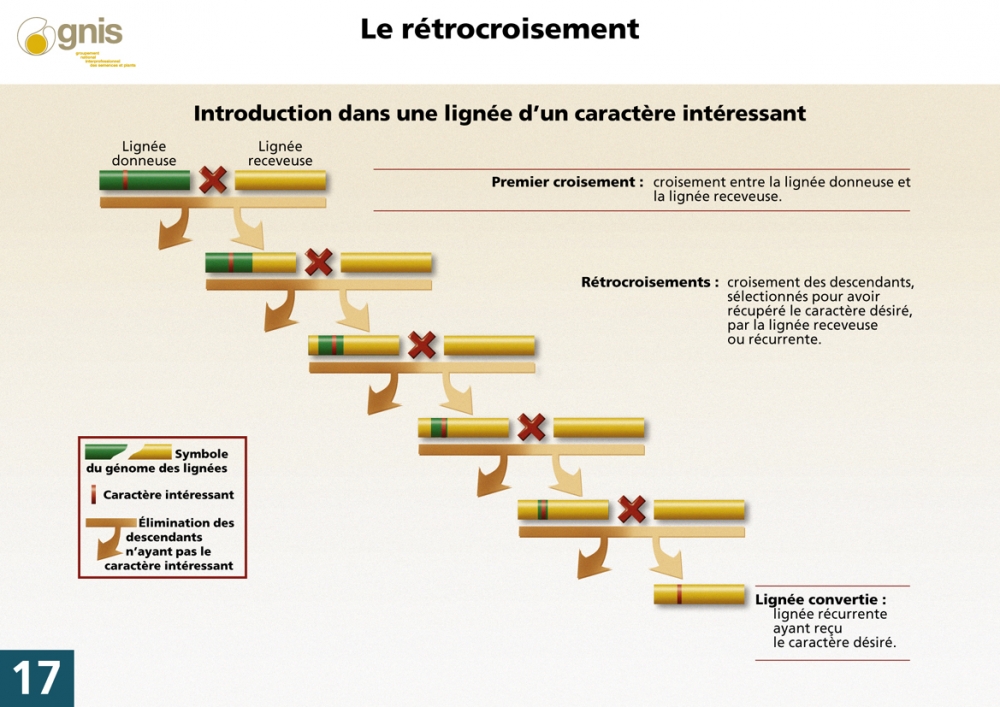
Figure 1 – Principe du rétrocroisement en sélection (source : GNIS pédagogie)
Quels sont les avantages de la SAM ?
La SAM permet de trier rapidement un grand nombre de plantes à un stade précoce du processus de sélection afin de recommencer rapidement un nouveau cycle de sélection (Figure 2). Cette méthodologie de sélection permet de cumuler le plus vite possible le maximum de gènes favorables dans un même génotype et ainsi de réduire considérablement le temps nécessaire à l’introduction de nouvelles variétés. Plusieurs années de travail de sélection peuvent ainsi être économisées pour chaque nouvelle variété produite.

Figure 2 : La SAM permet le tri d’un grand nombre de plants en sélection, à un stade de développement très précoce
En comparaison des méthodes de sélection traditionnelles, la SAM s’avère être :
- Plus rapide (quelques jours d’analyses au laboratoire suffisent pour établir un diagnostic de la présence ou de l’absence des gènes d’intérêt dans les plants en cours de sélection) ;
- Plus précise (détection fiable de la présence ou de l’absence du gène recherché) ;
- Non destructive car l’analyse des plants est réalisée sur une très faible quantité de matériel végétal (1 cm² de feuille, à titre d’exemple, Figure 3).
- Non influencée par des facteurs environnementaux.

Figure 3 : Prélèvement d’une très faible quantité de matériel végétale pour la réalisation des analyses de SAM au laboratoire.
La SAM présente cependant quelques limites. Elle est souvent inefficace pour la sélection de caractères agronomiques à déterminisme génétique complexe (comme le rendement, par exemple), gouvernés par un grand nombre de gènes ou de QTL (Quantitative Trait Loci) qui interagissent et dont la plupart sont encore inconnus.
Crédit photos : illustration plante/ADN : © Christos Georghiou – Fotolia.com ; Le rétrocroisement : © GNIS Pédagogie ; Photo tri de plants par SAM : © Vegenov-BBV ; Photo prélèvement de matériel végétal : © Vegenov-BBV





Laisser un commentaire